Álvaro Logullo Neto – alvarologullo@gmail.com
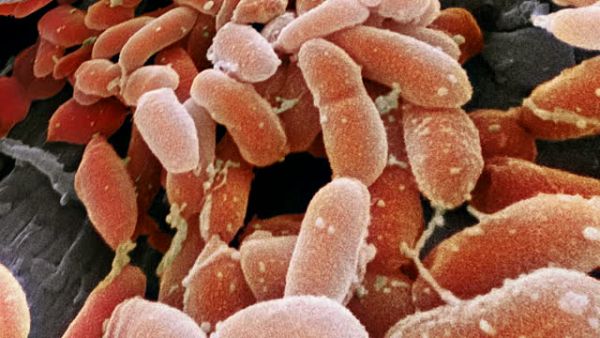
Pseudomonas aeruginosa é uma bactéria que pode ser encontrada em diversos ambientes, principalmente na água e no solo. Trata-se de um organismo patogênico oportunista, ou seja, não costuma provocar nenhum problema para indivíduos saudáveis. Entretanto, em pessoas com o sistema imunológico comprometido, a bactéria pode causar infecções. Alguns de seus alvos são pacientes com aids, vítimas de queimaduras e portadores de fibrose cística. A alta resistência de P. aeruginosa a antibióticos torna difícil o controle de infecções causadas por ela.
Regina Baldini, professora do Instituto de Química da USP, coordena um laboratório que investiga a fundo os mecanismos de funcionamento dessa bactéria: “O que a gente está estudando em nosso Laboratório são os sistemas, que chamamos sistemas de sinalização, que é a maneira como a bactéria sente um sinal do ambiente e transduz para dentro dela”. É sabido que uma população de bactérias que se encontra bem desenvolvida dentro de um indivíduo ativa genes chamados genes de virulência, produzindo enzimas que vão atacar o hospedeiro, quebrar a membrana de células e degradar proteínas. “Nosso trabalho é destrinchar esses sistemas de regulação, desde o momento em que bactéria sente o sinal até as proteínas que ativam ou reprimem a expressão de determinados genes”, conclui a professora.
Como explica Regina, para que a colônia se prolifere dentro do hospedeiro, as bactérias se utilizam de um mecanismo de comunicação conhecido como quorum sensing: “Trata-se de uma adaptação da bactéria para que no momento em que a população esteja pequena no paciente, o sistema imune não se preocupe com ela, porque ela está quieta. Depois que já está em uma população grande o suficiente começa a atacar o hospedeiro, aí o sistema imune já não dá mais conta”.
Após essa fase aguda de infecção, as bactérias, que já se encontram em abundância no paciente, passam a crescer na forma de biofilme. “Para formar um biofilme as bactérias se aderem em uma superfície, se juntam formando super colônias, perdem o flagelo, que é o que faz elas nadarem, e começam a produzir matrizes extracelulares, que são geralmente polissacarídeos, responsáveis por protegê-las”. Quando as bactérias se encontram “nadando” sozinhas no ambiente possuem um conjunto de genes e uma fisiologia completamente diferente de quando estão em um biofilme. “Isso dificulta o antibiótico penetrar e a maneira como elas estão fisiologicamente no biofilme também é uma maneira mais resistente a antibiótico do que quando elas estão soltas”, ressalta Regina.
Molécula c-di-GMP
Em busca de compreender melhor as interações que ocorrem dentro da célula, o Laboratório foca os estudos em uma molécula conhecida como c-di-GMP. Trata-se de um segundo mensageiro (pequena molécula de sinalização liberada pela célula e que provoca alterações fisiológicas na mesma) e que possui importante envolvimento na formação do biofilme. “Quando a bactéria tem altas concentrações dessa molécula [c-di-GMP], ela tende a formar biofilme, quando tem baixas concentrações, tende a viver sozinha”, explica Baldini.
As responsáveis por produzir c-di-GMP e por degradar essa molécula são enzimas diferentes. Sabendo que a bactéria P. aeruginosa possui muitas dessas enzimas dentro de sua célula, o Laboratório procura identificar as funções específicas exercidas por cada uma delas. Para tal, são realizados experimentos em laboratório, como descreve Regina: “A gente retira uma determinada proteína da célula e observa o que acontece com a bactéria. Será que ela deixou de fazer biofilme? Será que ela ficou mais sensível ao antibiótico? Outra estratégia é aumentar a concentração dessa proteína, o que a gente chama de super expressão”.
Proteína 72420
Pili (plural de pilus) ou fímbrias são estruturas presentes em bactérias e que se relacionam a sua capacidade de adesão na superfície: “Pseudomonas possui fímbrias em um polo só da célula. Tais fímbrias tem uma adesina na ponta, molécula que faz elas ligarem à superfície onde estão, algo muito importante para o começo da formação do biofilme”. Essas estruturas também servem para a bactéria se movimentar de uma maneira chamada twitching: “Ela gruda a adesina e encurta o pilus, depois estica de novo, semelhante uma lagarta mede palmo”, esclarece a professora.
Através de experiências em laboratório, o grupo de pesquisa identificou uma proteína específica (72420), responsável por produzir c-di-GMP e que interage com uma outra proteína (FimV) envolvida na montagem das fímbrias da bactéria.
Primeiramente, uma placa de sílica foi deixada no meio de cultura onde a bactéria cresce entre o meio e o ar. Assim que a bactéria grudou na placa, ela foi levada para análise em microscópio. Observou-se que a mutante com ausência da proteína 72420 cresce. Entretanto, as colônias de bactéria não conseguem se organizar para criar um biofilme. Em seguida, o gene dessa mesma proteína foi ligado ao gene da proteína verde luminescente, que é utilizada para uma melhor análise na microscopia, tornando mais visíveis as alterações. “Como a gente sabe que o pilus está no polo da bactéria e não em volta dela toda, a gente foi ver se a nossa proteína [72420] estava presente no polo ou não”, explica Regina. “O resultado foi positivo, pois observamos pontinhos brilhantes apenas nos polos das bactérias“.
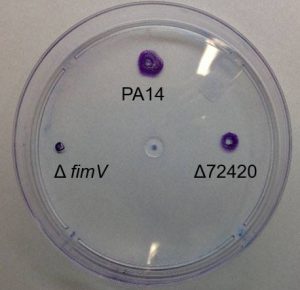
Uma proteína importante na montagem do pilus e que se localiza nos polos é FimV, que funciona como um arcabouço onde várias proteínas interagem. Foi possível observar, então, que as bactérias que não apresentam essa proteína FimV não formam mais pontinhos brilhantes de 72420, que se encontra espalhada por toda a célula. “Com isso a gente está mostrando que, como já sabíamos que a proteína FimV fica no polo, também sabemos agora que a nossa proteína depende dela [FimV] para ir para o polo também”. A conclusão é de que a proteína 72420 é responsável pelo aumento na quantidade de c-di-GMP em locais próximos aos pili das células, algo que favorece o crescimento do biofilme.
Perspectivas futuras
Regina Baldini também conta sobre outros modelos de pesquisa adotados em seu laboratório: “Fizemos uma experiência em camundongos: infectamos oito deles e aqueles que estavam com o gene selvagem morreram em dois dias, os com o gene mutante, chamado atvR, demoram mais para morrer e houve até um que sarou. Concluímos que esse gene é importante para a bactéria sobreviver durante a infecção.”
Tal conclusão foi alcançada após perceber-se que o mutante em AtvR estava sendo melhor combatido pelos macrófagos – células brancas do sangue utilizadas em experimentos em laboratório. Teoricamente, são os macrófagos que devem fagocitar as bactérias. Porém, no caso de P. aeruginosa, ela acabava por matar os macrófagos na grande maioria dos casos.
Ao longo dos últimos anos, o Laboratório tem buscado responder inúmeras perguntas relacionadas ao funcionamento da bactéria Pseudomonas aeruginosa: como o biofilme é formado? Quais proteínas interagem entre si? Como funcionam esses sinais? Qual a função específica de cada uma das proteínas na bactéria? Regina acredita que sua pesquisa e a de seus companheiros poderá responder todas elas no futuro, representando uma contribuição muito positiva para a ciência: “Apesar da nossa pesquisa ser muito básica, não estamos estudando um antibiótico novo, um tratamento novo, e sim, como a bactéria funciona, e também de que maneira ela consegue ficar mais resistente a antibióticos, pode ser que seja mais fácil desenvolver uma terapia contra ela no futuro, quando entendermos esses mecanismos”.



Faça um comentário